Revisión
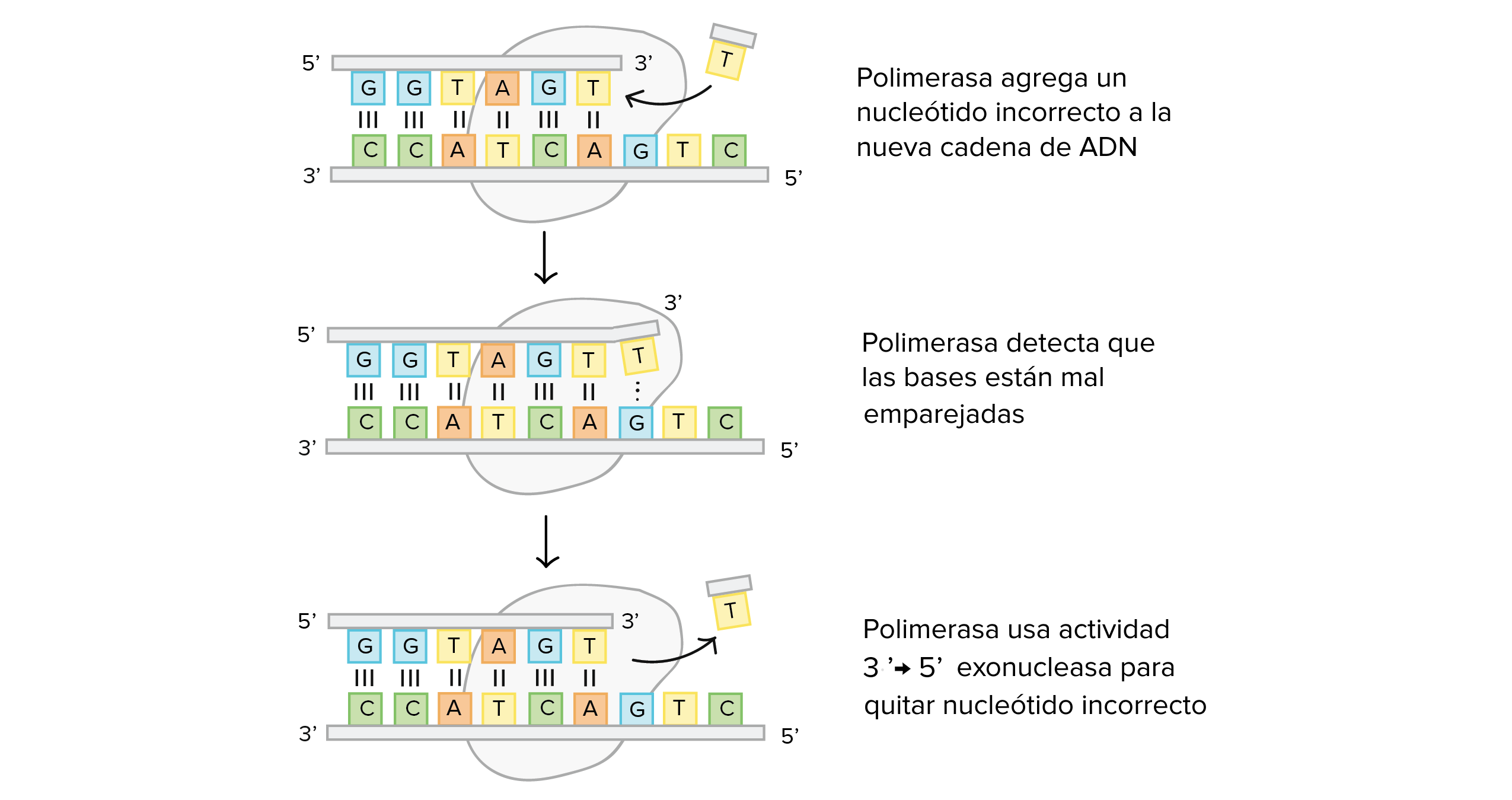
Las ADN polimerasas son las enzimas que forman el ADN en las células. Durante la replicación (copiado) del ADN, la mayoría de las ADN polimerasas pueden "revisar su trabajo" con cada base que añaden. Este proceso se llama revisión. Si la polimerasa detecta que ha agregado un nucleótido equivocado (apareado incorrectamente), lo quita y reemplaza enseguida, antes de continuar con la síntesis de ADN. (Berg, Tymoczko y Stryer, 2002).
Reparación de mal apareamiento
Muchos errores se corrigen con la revisión, pero algunos se escapan. La reparación de mal apareamiento sucede justo después de que se ha hecho ADN nuevo, y su función es eliminar y reemplazar las bases mal apareadas (las que no se arreglaron durante la revisión). La reparación de mal apareamiento también puede detectar y corregir pequeñas inserciones y deleciones que suceden cuando las polimerasas "se resbalan" y pierden su lugar sobre el molde. (Dexheimer, (2013).
squa¿Cómo funciona la reparación de mal apareamiento? Primero, un complejo proteico (grupo de proteínas) reconoce y se une a la base mal apareada. Un segundo complejo corta el ADN cerca de la pareja errónea y otras enzimas cortan el nucleótido incorrecto junto con un segmento de ADN circundante. Luego, una ADN polimerasa reemplaza la sección faltante con los nucleótidos correctos y una enzima llamada ADN ligasa sella el espacio.
Quizás te preguntes cómo es que las proteínas que participan en la reparación del ADN saben "qué está bien" durante la reparación de mal apareamiento. Es decir, cuando dos bases están mal apareadas (como G y T en el dibujo anterior), cuál de las dos se debe eliminar y reemplazar?
En bacterias, las cadenas de ADN originales y recién hechas se pueden diferenciar por una característica llamada estado de metilación. Una cadena de ADN antigua tiene grupos metilo en algunas de sus bases, mientras que sus cadenas de ADN recién hechas todavía no habrá recibido su grupo metilo. En eucariontes, los procesos que permiten identificar la cadena original en la reparación de mal apareamiento usan el reconocimiento de mellas (rupturas en las cadenas sencillas) presentes solo en el ADN recién sintetizado. (Cooper, 2000).
cube
Mecanismos de reparación de daños al ADN
En casi cualquier punto en la vida de una célula le pueden ocurrir cosas malas al ADN, no solo durante la replicación. De hecho, tu ADN se daña todo el tiempo por factores externos como la luz UV, productos químicos y los rayos X, ¡sin mencionar las reacciones químicas espontáneas que suceden incluso sin ofensas ambientales! (Dexheimer, 2013).
start superscript, 4, end superscriptAfortunadamente, tus células tienen mecanismos de reparación para detectar y corregir muchos tipos de daño al ADN. Los procesos de reparación que ayudan a arreglar el ADN dañado incluyen:
- Reversión directa: algunas reacciones químicas que dañan el ADN pueden ser "deshechas" directamente por enzimas de la célula.
- Reparación por escisión: el daño a una o unas cuantas bases de ADN se suele arreglar al eliminar (escindir) y reemplazar la región dañada. En la reparación por escisión de bases, solo se quita la base dañada. En la reparación por escisión de nucleótidos, como en la reparación de mal apareamiento que vimos antes, se elimina una sección de nucleótidos.
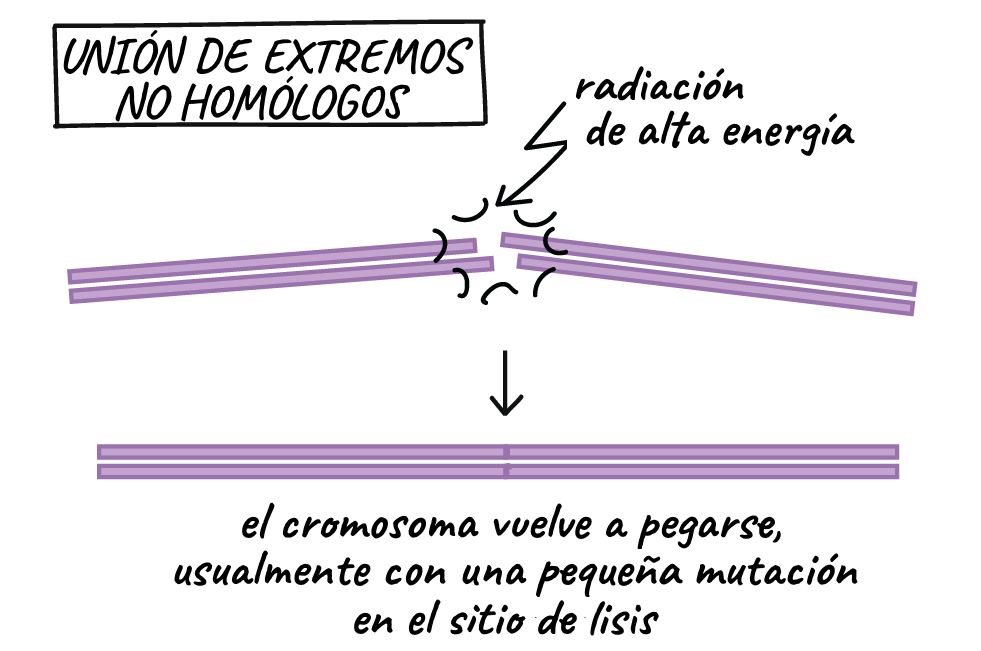
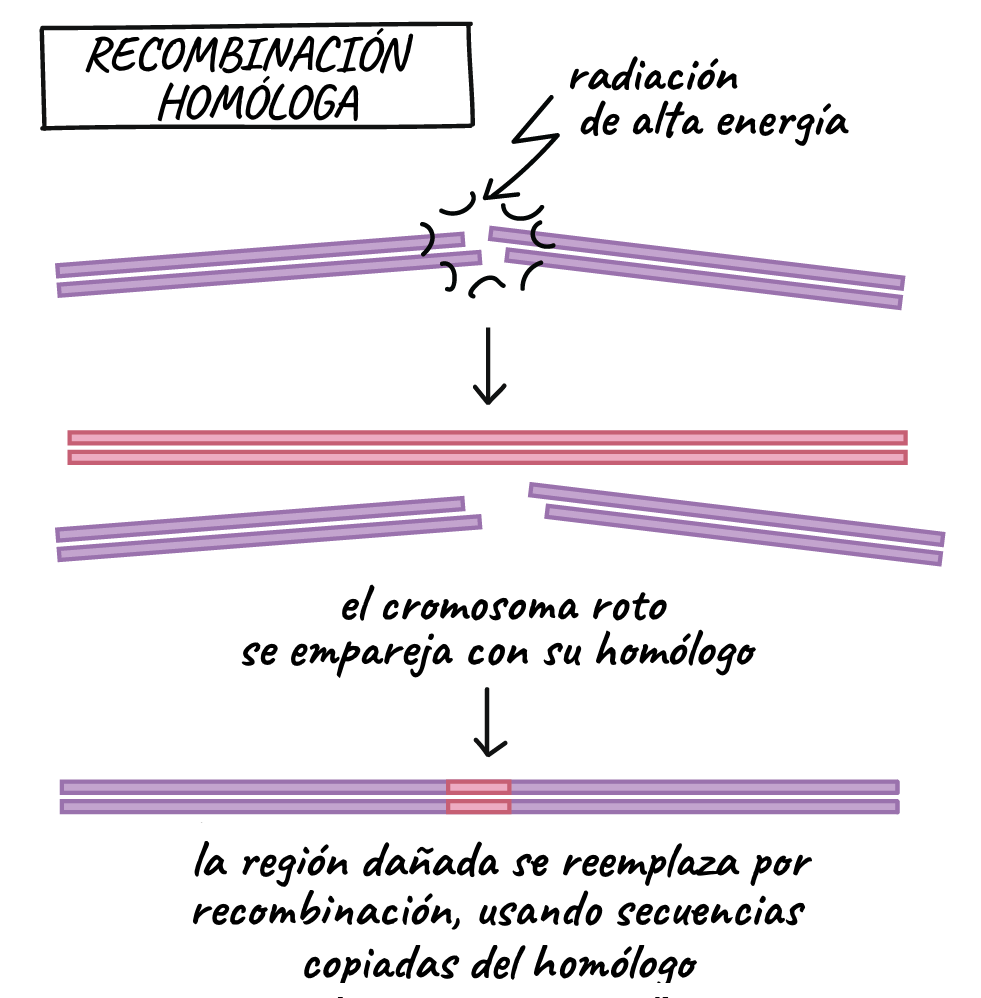
- Reparación de ruptura de la doble cadena: se utilizan dos vías principales, la unión de extremos no homólogos y la recombinación homóloga, para reparar rupturas en la doble cadena del ADN (es decir, cuando un cromosoma entero se divide en dos pedazos).
Reversión del daño
En algunos casos, una célula puede reparar daños en el ADN al simplemente revertir la reacción química que los causó. Para entender esto, necesitamos darnos cuenta que el "daño al ADN" suele implicar solo un grupo extra de átomos que se unen al ADN mediante una reacción química. Por ejemplo, la guanina (G) puede sufrir una reacción que añade un grupo metilo a un átomo de oxígeno de la base. Si no se corrige, la guanina que contiene el metilo formará pareja con una timina (T) en lugar de la citocina (C) durante la replicación del ADN. Afortunadamente, los seres humanos y muchos otros organismos tienen una enzima que puede quitar el grupo metilo, y revertir la reacción para regresar la base a su estado normal. (Cooper, 2000).
Reparación por escisión de base
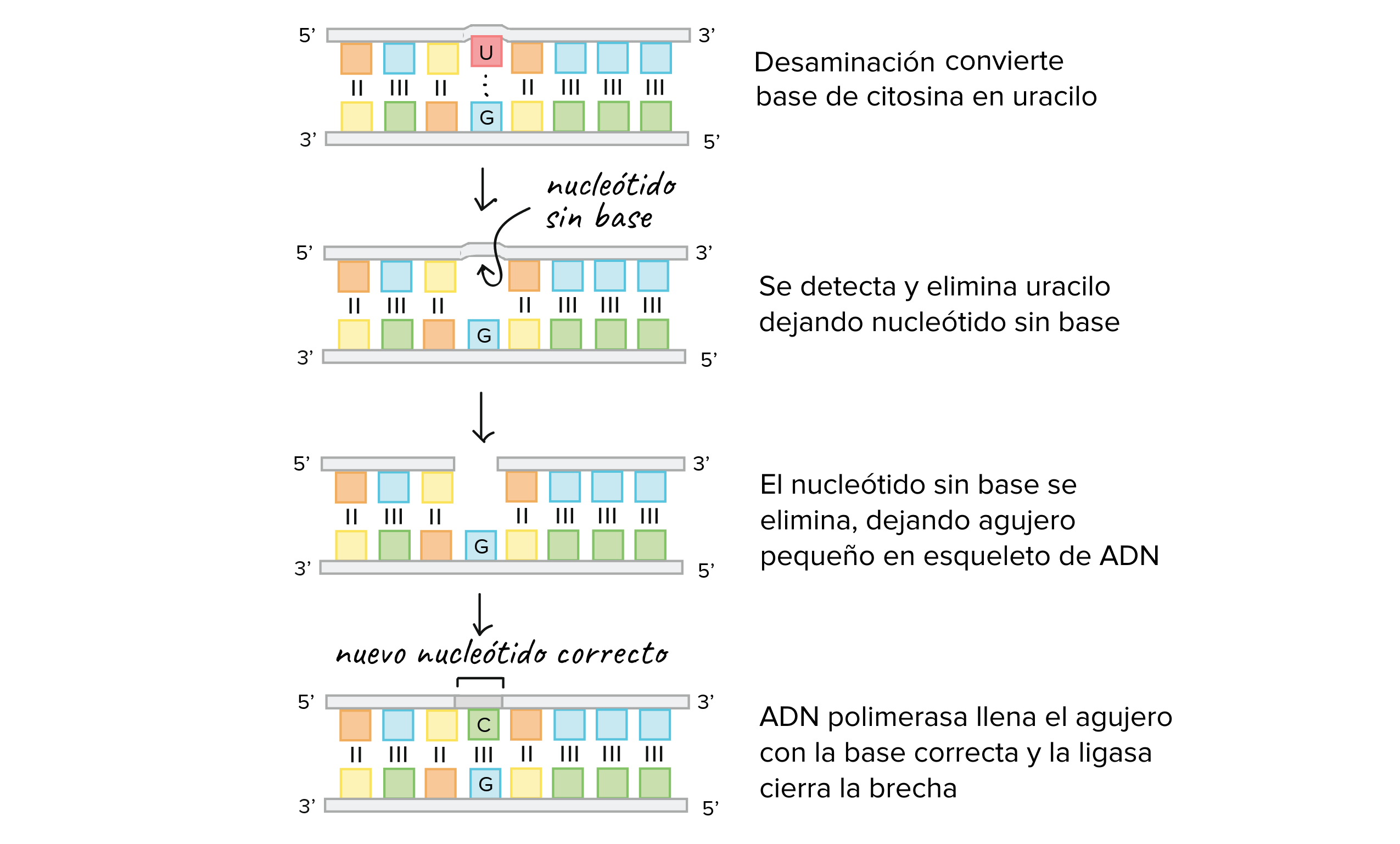
La reparación por escisión de base es un mecanismo que se usa para detectar y eliminar ciertos tipos de bases dañadas. Un grupo de enzimas llamadas glicosilasas tiene un papel clave en la reparación por escisión de bases. Cada glicosilasa detecta y elimina un tipo específico de base dañada.
Por ejemplo, una reacción química llamada desaminación puede convertir una base citosina en uracilo, una base que suele encontrarse solo en el ARN. Durante la replicación del ADN, el uracilo formará pareja con adenina en lugar de guanina (como lo haría si la base todavía fuera citosina), por lo que un intercambio de citosina por uracilo sin corregir puede causar una mutación. (Cooper, 2000).
start superscript, 5, end superscripReparación por escisión de nucleótidos
La reparación por escisión de nucleótidos es otra vía que se usa para eliminar y reemplazar bases dañadas. La reparación por escisión de nucleótidos detecta y corrige tipos de daño que distorsionan la doble hélice del ADN. Por ejemplo, esta vía detecta bases que han sido modificadas con grupos químicos voluminosos, como los que se unen a tu ADN cuando se expone a las sustancias químicas del humo de cigarrillos (Dexheimer, 2013).
La reparación por escisión de nucleótidos también se utiliza para reparar algún tipo de daño que causa la radiación UV, como cuando te quemas con el sol. La radiación UV puede causar que la citosina y la timina reaccionen con bases vecinas que también sean C y T, y se forman enlaces que distorsionan la doble hélice y causan errores en la replicación del ADN. El tipo más común de unión, el dímero de timina, se compone de dos bases de timina que reaccionan entre sí y se unen químicamente (Goodsell, 2007).
En la reparación por escisión de nucleótidos se elimina el nucleótido (o nucleótidos) con daño junto con un segmento circundante de ADN. En este proceso una helicasa (enzima que abre el ADN) abre el ADN para formar una burbuja y enzimas que cortan el ADN quitan el segmento dañado de la burbuja. Una ADN polimerasa reemplaza el ADN que falta y una ADN ligasa sella la brecha en el esqueleto de la cadena (Dexheimer, 2013).
start superscript, 9, end superscriptReparación de rotura de la doble cadena
Algunos factores ambientales, como la radiación de alta energía, pueden causar roturas en la doble cadena del ADN (rompen un cromosoma en dos). Este es el tipo de daño al ADN que se relaciona con las historias del origen de los superhéroes en los cómics y con desastres como Chernobyl en la vida real.
Las roturas de la doble cadena son peligrosas porque pueden perderse grandes segmentos de cromosomas y los cientos de genes que contienen si la fragmentación no se repara. Dos vías que participan en la reparación de rotura del ADN de doble cadena son la vía de unión de extremos no homólogos y la de recombinación homóloga.
En la unión de extremos no homólogos, los dos extremos rotos de un cromosoma simplemente se vuelven a pegar. Este mecanismo de reparación es "desordenado" y por lo general resulta en la pérdida, o a veces adición, de unos cuantos nucleótidos en el sitio de corte. Por lo tanto, la unión de extremos no homólogos tiende a producir una mutación, pero eso es mejor que la alternativa (la pérdida de un brazo entero del cromosoma). (Albert, Johnson Lewis, Raff, Roberts y Walter, 2002).
En la recombinación homóloga, se utiliza la información del cromosoma homólogo que coincide con la del dañado (o de una cromátida hermana si el ADN se ha copiado) para reparar la fragmentación. En este proceso se acercan los dos cromosomas homólogos y se utiliza la región sin daños del homólogo o la cromátida como molde para sustituir la región dañada del cromosoma roto. La recombinación homóloga es "más limpia" que la unión de extremos no homólogos y no suele causar mutaciones (Kimball, 2015).
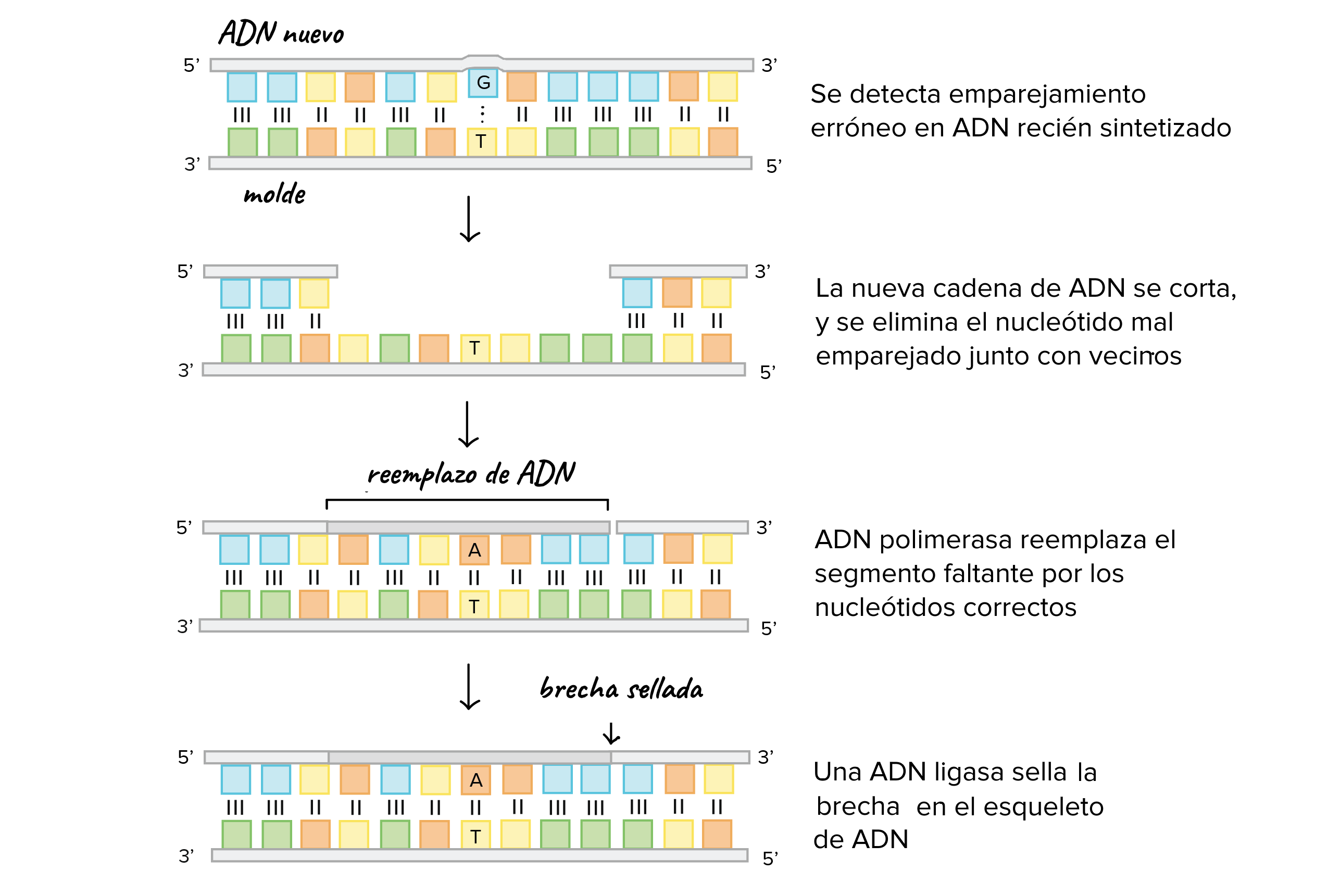

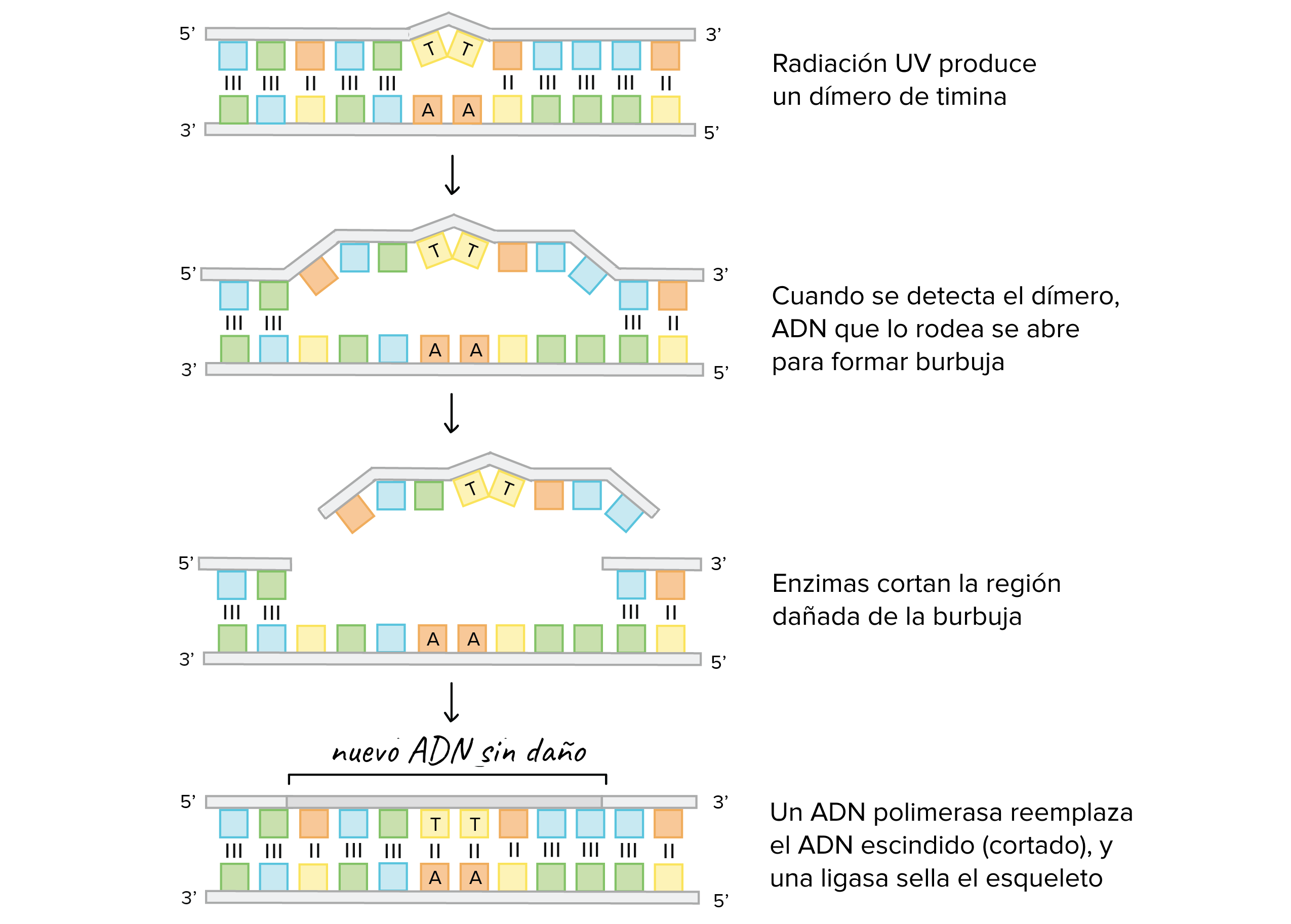
Comentarios
Publicar un comentario